BMW to recall 44,052 cars with safety hazards in China
Дезоксирибонуклейны х?чил (ДНХ) нь мэдэгдэж буй б?х амьд организмуудын ?с?лт х?гжилт, ажиллагаанд ашиглагддаг ба генетикийн заавруудыг агуулсан нуклейны х?чил юм. ДНХ-ийн молекулуудын гол ??рэг нь удаан хугацаанд мэдээллийг хадгалдаг ба энэ нь эсийн б?рэлдэх??н хэсг??дийг тухайлбал уургууд болон Рибонуклейн х?члийн молекулуудыг угсрах заавруудыг агуулдаг учираас ДНХ-г ихэвчлэн барилгын план зургуудын цуглуулгатай адилтгадаг. Генетикийн энэх?? мэдээллийг агуулдаг ДНХ-ийн хэсг??дийг ген гэдэг. Гэхдээ бусад ДНХ-ийн дарааллууд нь б?тцийн зориулалттай байдаг эсвэл энэх?? генетикийн мэдээллийг хэрхэн ашиглахыг зохицуулахад хэрэглэгддэг.
Химийн хувьд ДНХ нь нуклеотид хэмээх энгийн нэгж??дээс б?рдсэн, диэфирийн холбоогоор холбогдсон чихэр болон фосфатын нэгдлээс б?рдсэн нуруу б?хий хоёр урт полимероос тогтдог. Эдгээр хоёр хэлхээ нь бие биетэйгээ параллел боловч чиглэл нь эсрэг оршидог. Чихэр б?рт д?рв?н т?рлийн молекулуудын нэг нь холбогдсон байх ба эдгээрийг сууриуд гэдэг. Гол нурууны дагуу байрлах эдгээр д?рв?н суурийн дараалал нь мэдээллийг кодлодог. Энэ мэдээллийг генетик кодыг ашиглан уншдаг. Генетик код нь протейнуудад байх амин х?чилийн дараалалыг тодорхойлж ?гд?г. Кодыг ДНХ-ийн хэсг??дийг холбогдох нуклейны х?чил РНХ-р?? хуулбарлах замаар уншдаг. Энэ процессыг генетик х?рв??лэлт гэдэг. Тэдгээр РНХ молекулуудын ихэнх нь протейныг нийлэгж??лэхэд ашиглагддаг ба харин бусад нь б?тэц??д болох жишээлбэл рибосом болон сплицезонд шууд ашиглагддаг.
Эсийн дотор ДНХ нь хромосом гэгдэх б?тц??дэд хуваагдах ба эсийн доторх хромосомуудын нэгдэл нь геномыг б?тээдэг. Тэдгээр хромосомууд нь эсийн хуваагдалын ?мн? хуулбарлан хуваагддаг. Энэ процессыг ДНХ ?ржих гэдэг. Эукариот организмууд тухайлбал амьтан, ургамал, м??г зэрэг нь ??рсдийн ДНХ-г эсийн б??м дотороо хадгалдаг бол харин Прокариот организмууд тухайлбал бактериийх эсийн цитоплазм дотороо байдаг. Хромосомуудын дотор хроматин протейнууд тухайлбал гистонууд ДНХ-г нягтруулж, зохион байгуулдаг. Энэ нь бусад протейнуудтай харилцан ?йлчлэлцэхийг хянахад тусладаг б?г??д улмаар генийн х?рв??лэлтийг хянадаг.
Физик химийн шинж чанар
[засварлах | кодоор засварлах]
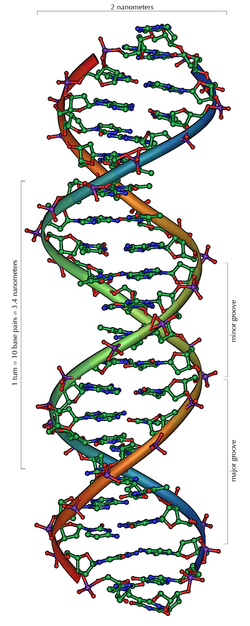
ДНХ нь нуклеотидууд гэж нэрлэгдэх дахин давтагдах нэгж??дээс б?рдсэн урт полимер юм.[1][2] ДНХ-ийн гинж нь 22-оос 26 Ангстром ?рг?н (2.2-оос 2.6 нанометр) б?г??д нэг нуклеотид нь 3.3 Ангстром (0.33 нанометр) урт байдаг.[3] Давтагдах нэгж??д нь хэдий маш жижигхэн ч гэсэн ДНХ-ийн полимерууд нь хэдэн сая нуклеотидуудыг агуулсан асар том молекулууд байж болдог. Тухайлбал х?ний хамгийн том хромосом болох Хромосом № 1 нь 220 сая хос сууриас б?рдсэн урт байдаг.[4]
Амьд организмуудад ДНХ нь ганц молекулын байдлаар байнга оршдогг?й, харин нарийн холбогдсон хос молекулууд байдлаар оршдог.[5][6] Тэдгээр хоёр урт хэлхээ нь м?лх?? ургамал адил с?лжилдэн хос спираль хэлбэрийг олдог. Нуклеотидын давтамж нь гинжийг холбон барих молекулын нурууны хэсгийг болон спираль дах бусад ДНХ-ийн хэлхээтэй харилцан ?йлчлэх суурийг хоюуланг нь агуулдаг. Ер?нхийд?? сахарт холбогдсон суурийг нуклеозид гэдэг ба сахарт болон нэг эсвэл хэд хэдэн фосфатын нэгдэлд холбогдсон суурийг нуклеотид гэдэг. Хэрэв олон нуклеотидууд хоорондоо ДНХ-д байдаг шиг холбогдсон байвал энэ полимерийг полинуклеотид хэмээн дурддаг.[7]
ДНХ-ийн хэлхээний гол нуруу нь фосфат болон сарартийн тунадаснуудын хослолоос б?рддэг.[8] ДНХ дэхь сахар нь 2-дезоксироз ба энэ нь пентоз (таван н??рст?р?гч) сахар юм. сахарууд нь хоорондоо фосфатын нэгдэлээр холбогдоно. Фосфатын нэгдэл нь сахарын 3-р болон 5-р орбитын атомуудын хооронд фосфодиэфирийн холбоосыг ??сгэдэг. Эдгээр тэгш биш хэмт холбоосууд нь ДНХ-ийн хэлхээ нь чиглэлтэй болохыг харуулж байгаа юм. ДНХ-ийн хос спираль дахь нэг хэлхээний нуклеотидуудын чиглэл нь н?г?? хэлхээнийхээ нуклеотидуудын эсрэг чиглэлд байдаг. Энэх?? ДНХ-ийн хэлхээн??дийн тогтоцыг антипараллел гэдэг. ДНХ-ийн хэлхээн??дийн тэгш биш хэмт т?гсг?л??дийг 5′ (таван штрих) болон 3' (гурван штрих)-ын т?гсг?л??д гэж нэрлэдэг. ДНХ болон РНХ хоёрын хамгийн гол ялгаа нь сахар б?г??д 2-дезоксиробозын оронд ??р нэгэн сахар болох рибоз нь РНХ-д байдаг.[6]
ДНХ-ийн хоёр хэлхээнд буй сууриудыг хооронд нь холбосон уст?р?гчийн холбоосууд нь ДНХ-ийн хос спиралийг тогтворжуулах ??ргийг г?йцэтгэдэг. ДНХ дэхь д?рв?н суурийг аденин (A), цитозин (Ц), гуанин (Г), тимин (Т) гэдэг. Доор харуусан эдгээр д?рв?н сууриуд нь чихэр/фосфатад холбогдон аденозин монофосфатад харуулсаны дагуу б?тэн нуклеотидыг ??сгэдэг.
Эдгээр сууриудыг хоёр т?рлийн ангилалд хуваадаг. Аденин болон гуанинууд нь пурин гэгддэг 5-р болон 6-р т?вшинд нэгдсэн гетеро цагирагт нэгдэл??д юм. Харин цитозин болон тимин нь пиримидин гэгдэх 6-р т?вшиний орбитын нэгдэл юм.[6] 5 дахь пиримидин суурийг урацил (У) гэх ба энэ нь ихэвчлэн РНХ-д тиминий ??ргийг орлох б?г??д тиминээс орбит дээрээ метилийн б?лэгг?йгээрээ ялгардаг. Урацил нь ДНХ-д бараг илэрдэгг?й б?г??д з?вх?н цитозин задрах ?ед илэрдэг. Гэхдээ ??нээс гадна з?вх?н PBS1 гэж нэрлэгддэг фаг нь ??рийнх?? ДНХ-д урацилыг агуулж байдаг.[9] Дээрхээс ялгаатай нь дараах РНХ-ийн тодорхой молекулуудын нийлэгжилт юм. Энэ нь байхг?й метилийн б?лгийн ферментийн нэмэлтээр ихээхэн хэмжээний урацилууд тимин??д болон хувирах нийлэгжилт юм. Энэ нь ихэвчлэн шилжих РНХ болон рибозон РНХ мэтийн б?тцийн болон ферментийн РНХ-??дэд илэрдэг.[10]
Их болон бага ховил
[засварлах | кодоор засварлах]
Хос спирал нь баруун гарын д?рмээр эргэх спирал юм. ДНХ-ийн хэлхээн??д бие биеээ тойрч эргэх ?ед фосфат нуруунуудын хооронд зай ??сэх б?г??д доторх сууриудын талуудыг ил гарган харуулна (амьлуулсан д?рслэлийг харна уу). Хос спиралын гадаргуу дээр мушгиралдсан хоёр янзын ховил байна. Нэг ховилыг нь их ховил гэх б?г??д 22 ? ?рг?нтэй, харин н?г??г нь бага ховил гэх б?г??д 12 ? ?рг?нтэй байдаг.[12] Бага ховил нь нарийн байгаа нь сууриудын ирмэг??д нь их ховилын талаасаа ил?? нээлттэй байна гэсэн ?г юм. ??ний ?р д?нд давхар хэлхээ б?хий ДНХ-ийн тодорхой дараалалд барьцалдсан активаторууд мэтийн протейнууд нь их ховилын талаасаа ил гарсан сууриудтай холболт ??сгэдэг.[13]
Сууриудын хослол
[засварлах | кодоор засварлах]
|

|
Нэг талын хэлхээнд буй суурь б?р нь н?г?? хэлхээнд буй суурь б?ртэй холбоос ??сгэдэг. ??нийг сууриудын хослол ??сгэх гэдэг. Энд пуринууд нь пиримидин??дтэй уст?р?гчийн холбоог ??сгэн А нь з?вх?н Т-тэй, Ц нь з?вх?н Г-тэй холбогдох замаар хоорондоо холбогддог. Хос спиралын дагуу хоорондоо ийнх?? барьцалдсан хоёр нуклеотидуудын тогтоцыг суурийн хослол гэж нэрлэдэг. Хос спиралын хоёр хэлхээ нь ДНХ-ийн дараалалын н?л??л?л??с ангид байх пи холбоо болон гидрофобын эффектээс ??сэх х?чний ?йлчлэлээр м?н хоорондоо барьцалддаг.[14] Уст?р?гчийн холбоонууд нь ковалент биш учир задрах болон буцаж холбогдохдоо амархан байдаг. Тиймээс ДНХ-ийн хос спирал дахь хоёр хэлхээг механик х?ч эсвэл ?нд?р температурын н?л??г??р ?мдний цахилгааныг тайлж буй мэт салгаж болдог.[15] ??ний ?р д?нд ДНХ-ийн спиралын хос хэлхээт дараалал дахь б?х мэдээлэл нь хэлхээ тус б?ртээ хуулбарлагддаг. Энэ онцлог нь ДНХ-ийн ?ржилд чухал ??рэг г?йцэтгэдэг юм. ?нэндээ суурийн хослолын энэх?? давтагдах чанартай, онцгой дотоод харилцан ?йлчлэл нь амьд организмуудын ДНХ-ийн б?хий л ажиллагаанд нэн чухал байдаг билээ.[1]
Хоёр т?рлийн суурийн хослолууд нь ??р ??р тоотой уст?р?гчийн холбоонуудыг ??сгэдэг. АТ нь уст?р?гчийн хоёр холбоог, харин ГЦ нь уст?р?гчийн гурван холбоог ??сгэдэг (з??н талын зургийг харна уу). Тиймээс ГЦ суурийн хослол нь АТ суурийн хослолоос ил?? бат бэх байдаг. ??р??р хэлбэл ДНХ-ийн хоёр хэлхээний хоорондын харилцан ?йлчлэлийн хэмжээ нь ДНХ-ийн хос спиралын нийт урт болон т??нд агуулагдах ГЦ суурийн хослолын эзлэх хувиас шууд хамаарна. ГЦ хослолын агуулга ?нд?ртэй, урт ДНХ-ийн спирал нь х?чтэй харилцан ?йчлэл б?хий хэлхээн??дтэй байдаг ба харин АТ хослолын агуулга ?нд?ртэй, богино ДНХ-ийн хувьд сул харилцан ?йлчлэл б?хий хэлхээн??дтэй байна.[16]
Амархан салгагддаг ДНХ-ийн хос спиралын хэсг??дэд тухайлбайл бактерийн промоторуудад байдаг ТАТААТ Прибнов хайрцагт АТ хослолын агуулга ?нд?р байх хандлага ажиглагддаг б?г??д энэ нь хэлхээн??дийг салгахад амархан болгодог.[17] Лабораторийн горимд энэх?? харилцан ?йлчлэлийн х?чний хэмжээг уст?р?гчийн холбоонууд нь задрахад шаардлагатай температурын хэмжээгээр тодорхойлдог. ??р??р хэлбэл тэдгээрийн хайлах температур (м?н Tm утга ч гэж хэлдэг ) гэж нэрлэдэг. ДНХ-ийн хос спиралын б?х суурийн хослолууд хайлж дуусах ?ед хэлхээн??д нь салах б?г??д тэдгээрийг уусмалд хоёр огт хамааралг?й молекулууд байдалаар байлгаж болдог. Эдгээр ганц хэлхээт ДНХ-ийн молекулуудад тодорхой нийтлэг хэлбэр д?рс байдагг?й б?г??д харин зарим конформацууд нь харьцангуй тогвортой байдаг.[18]
Эерэг ба с?р?г туйл
[засварлах | кодоор засварлах]Хэрэв ДНХ-ийн дараалал нь протейнд х?рв??лэгдсэн зуучлагч РНХ-ийн хуулбарын дараалалтай ижил байвал ??нийг "туйлт" (эерэг туйлт) дараалал гэдэг. Харин эсрэг хэлхээний дараалал нь туйлт дараалалтай хосмог ??сгэж байвал т??нийг "антитуйлт" буюу с?р?г туйлт дараалал гэнэ. Нэгэнт РНХ-ийн полимеразууд нь ??рийнх?? загваруудын хосмог хуулбарыг ??сгэх замаар явдаг тул с?р?г туйлт хэлхээ нь эерэг туйлт зуучлагч РНХ-г ??сгэх загвар болдог. Эерэг болон с?р?г туйлт дараалалууд нь хоёул ДНХ-ийн нэг хэлхээний ??р ??р хэсг??д дээр орших боломжтой (тухайлбал хоёр хэлхээ нь хоёул эерэг, с?р?г дараалалыг агуулж байх тохиолдолд). Прокариот эукариот хоёуланд нь с?р?г туйлт РНХ-ийн дараалал б?рэлддэг боловч эдгээр РНХ-ийн ажиллагаа нь б?хэлдээ тодорхой биш юм.[19] Нэг тайлбар байдаг б?г??д ??нд с?р?г туйлт РНХ-??д нь РНХ-РНХ суурийн хослолын дагуу генийн илэрхийлэлийг зохицуулахад оролцдог гэсэн байдаг.[20]
Прокариот болон эукариотуудад олддог ц??х?н хэдэн, харин плазмид болон вирус??дэд т?гээмэл олддог ДНХ-ийн дараалалууд нь гени?д нь давхарддаг улмаас дээр гаргасан эерэг болон с?р?г туйлт хэлхээн??дийн ялгааг б?дгэр??лдэг.[21] Эдгээр тохиолдолуудад зарим ДНХ-ийн дараалалууд нь давхар ??рэгтэй оролцдог б?г??д тухайлбал нэг хэлхээний дагуух 5' болон 3'-уудыг унших ?ед нэг протейн нь кодлодог бол н?г?? протейн нь н?г?? хэлхээний дагуу эсрэг чиглэлд унших ?ед кодлодог. Бактериудад энэх?? давхцал нь магадг?й генийн бичиглэлийг зохицуулахад ашиглагддаг байж болох юм.[22] Харин вир?с??дэд давхардсан гени?д нь вир?сийн жижиг геномд кодлогдсон мэдээлэлийн хэмжээг ихэсгэдэг.[23] Геномын хэмжээг багасгах ??р замыг шугаман эсвэл дугариг дан хэлхээт ДНХ-г генетикийн материал болгон ашигладаг зарим вир?с??д дээр харж болох юм.[24][25]
Супер-ороомог
[засварлах | кодоор засварлах]ДНХ-г утас ээрэхтэй адилаар мушгих процессыг ДНХ-г ээрэх гэдэг. ДНХ нь "тайван" т?л?вт орших ?ед хэлхээ нь хос спиралын тэнхлэгийг 10.4 суурийн хослол тутамд нэг б?тэн ороодог. Гэхдээ ДНХ-г ээрвэл хэлхээн??д нь ил?? чанга эс б?г??с ил?? сул болно.[26] Хэрэв ДНХ-г спиралын чиглэлийн дагуу ээрвэл ??нийг эерэг супер-ээр??лдэх гэх б?г??д сууриуд нь хоорондоо ил?? шиг?? барьцалдана. Харин эсрэг чиглэлд ээрэх тохиолдолд энэ нь с?р?г супер-ээр??лдэх болох б?г??д сууриуд нь ил?? салангид болно. Байгаль дээр ихэнх ДНХ-г топоизомеразууд гэгдэх фермент??дийн тусламжтайгаар бага зэрэг с?р?г супер-ээр??лддэг.[27] Эдгээр фермент??д нь х?рв??лэлт болон ДНХ ?рж??лэх процессуудад ДНХ-ийн хэлхээн??дийг ээрэх зорилгоор ашиглагддаг.[28]

Бусад давхар-мушгиа б?тэц??д
[засварлах | кодоор засварлах]ДНХ нь хэд хэдэн боломжит конформац хэлбэрт оршдог. Гэвч з?вх?н A-ДНХ, B-ДНХ болон Z-ДНХ-г л байгаль дээр буй биологийн систем??дэд ажиглаж болно. ДНХ нь ямар конформацтай байх нь ДНХ-ийн дараалал, т??ний супер-ороомогийн хэмжээ болон чиглэл, сууриудын химийн ??рчл?лт, м?н уусмалын н?хц?л тухайлбал метал ионууд болон полиаминууд зэрэгээс хамаардаг.[29] Эдгээр гурван конформацуудаас дээр дурдагдсан "В" хэлбэр нь эс??дийн дотороос олддог орчинд хамгийн т?гээмэл байдаг.[30] Бусад хоёр ДНХ-ийн хос-мушгиа хэлбэр??д нь геометр д?рс болон чиглэл??дээрээ ялгагддаг.
"А" хэлбэр нь ил?? ?рг?н, баруун эрчилсэн спираль б?г??д т??ний бага ховил нь г?ехэн болоод ?рг?н, харин их ховил нь г?нзгий болоод нарийн байдаг. "А" хэлбэр нь ДНХ-ийн дегидратацад орсон физиологийн бус н?хц?лд илэрдэг б?г??д харин эсийн дотор ДНХ болон РНХ-ийн хэлхээн??дийн холимог хослолоос, м?н фермент-ДНХ комлексоос ??сэх магадлалтай.[31][32]
М?н ?зэх
[засварлах | кодоор засварлах]Ишлэл
[засварлах | кодоор засварлах]- ↑ 1.0 1.1 Alberts, Bruce (2002). Molecular Biology of the Cell; Fourth Edition. New York and London: Garland Science. ISBN 0-8153-3218-1.
{{cite book}}: Unknown parameter|coauthors=ignored (|author=suggested) (help) - ↑ Butler, John M. (2001) Forensic DNA Typing "Elsevier". pp. 14–15. ISBN 978-0-12-147951-0.
- ↑ Mandelkern M, Elias J, Eden D, Crothers D (1981). "The dimensions of DNA in solution". J Mol Biol. 152 (1): 153–61. PMID 7338906.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Gregory S; et al. (2006). "The DNA sequence and biological annotation of human chromosome 1". Nature. 441 (7091): 315–21. PMID 16710414.
{{cite journal}}: Explicit use of et al. in:|author=(help) - ↑ Watson J, Crick F (1953). "Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid" (PDF). Nature. 171 (4356): 737–8. PMID 13054692.
- ↑ 6.0 6.1 6.2 Berg J., Tymoczko J. and Stryer L. (2002) Biochemistry. W. H. Freeman and Company ISBN 0-7167-4955-6
- ↑ Abbreviations and Symbols for Nucleic Acids, Polynucleotides and their Constituents IUPAC-IUB Commission on Biochemical Nomenclature (CBN), Accessed 03 Jan 2006
- ↑ Ghosh A, Bansal M (2003). "A glossary of DNA structures from A to Z". Acta Crystallogr D Biol Crystallogr. 59 (Pt 4): 620–6. PMID 12657780.
- ↑ Takahashi I, Marmur J. (1963). "Replacement of thymidylic acid by deoxyuridylic acid in the deoxyribonucleic acid of a transducing phage for Bacillus subtilis". Nature. 197: 794–5. PMID 13980287.
- ↑ Agris P (2004). "Decoding the genome: a modified view". Nucleic Acids Res. 32 (1): 223–38. PMID 14715921.[permanent dead link]
- ↑ Created from PDB 1D65
- ↑ Wing R, Drew H, Takano T, Broka C, Tanaka S, Itakura K, Dickerson R (1980). "Crystal structure analysis of a complete turn of B-DNA". Nature. 287 (5784): 755–8. PMID 7432492.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Pabo C, Sauer R (1984). "Protein-DNA recognition". Annu Rev Biochem. 53: 293–321. PMID 6236744.
- ↑ Ponnuswamy P, Gromiha M (1994). "On the conformational stability of oligonucleotide duplexes and tRNA molecules". J Theor Biol. 169 (4): 419–32. PMID 7526075.
- ↑ Clausen-Schaumann H, Rief M, Tolksdorf C, Gaub H (2000). "Mechanical stability of single DNA molecules" (PDF). Biophys J. 78 (4): 1997–2007. PMID 10733978. Эх хувилбараас архивласан: 2025-08-06. Татаж авсан: 2025-08-06.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Chalikian T, V?lker J, Plum G, Breslauer K (1999). "A more unified picture for the thermodynamics of nucleic acid duplex melting: a characterization by calorimetric and volumetric techniques" (PDF). Proc Natl Acad Sci U S A. 96 (14): 7853–8. PMID 10393911. Эх хувилбараас архивласан: 2025-08-06. Татаж авсан: 2025-08-06.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ deHaseth P, Helmann J (1995). "Open complex formation by Escherichia coli RNA polymerase: the mechanism of polymerase-induced strand separation of double helical DNA". Mol Microbiol. 16 (5): 817–24. PMID 7476180.
- ↑ Isaksson J, Acharya S, Barman J, Cheruku P, Chattopadhyaya J (2004). "Single-stranded adenine-rich DNA and RNA retain structural characteristics of their respective double-stranded conformations and show directional differences in stacking pattern". Biochemistry. 43 (51): 15996–6010. PMID 15609994.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Hüttenhofer A, Schattner P, Polacek N (2005). "Non-coding RNAs: hope or hype?". Trends Genet. 21 (5): 289–97. PMID 15851066.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Munroe S (2004). "Diversity of antisense regulation in eukaryotes: multiple mechanisms, emerging patterns". J Cell Biochem. 93 (4): 664–71. PMID 15389973.
- ↑ Makalowska I, Lin C, Makalowski W (2005). "Overlapping genes in vertebrate genomes". Comput Biol Chem. 29 (1): 1–12. PMID 15680581.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Johnson Z, Chisholm S (2004). "Properties of overlapping genes are conserved across microbial genomes". Genome Res. 14 (11): 2268–72. PMID 15520290.
- ↑ Lamb R, Horvath C (1991). "Diversity of coding strategies in influenza viruses". Trends Genet. 7 (8): 261–6. PMID 1771674.
- ↑ Davies J, Stanley J (1989). "Geminivirus genes and vectors". Trends Genet. 5 (3): 77–81. PMID 2660364.
- ↑ Berns K (1990). "Parvovirus replication". Microbiol Rev. 54 (3): 316–29. PMID 2215424.
- ↑ Benham C, Mielke S (2005). "DNA mechanics". Annu Rev Biomed Eng. 7: 21–53. PMID 16004565.
- ↑ Champoux J (2001). "DNA topoisomerases: structure, function, and mechanism". Annu Rev Biochem. 70: 369–413. PMID 11395412.
- ↑ Wang J (2002). "Cellular roles of DNA topoisomerases: a molecular perspective". Nat Rev Mol Cell Biol. 3 (6): 430–40. PMID 12042765.
- ↑ Basu H, Feuerstein B, Zarling D, Shafer R, Marton L (1988). "Recognition of Z-RNA and Z-DNA determinants by polyamines in solution: experimental and theoretical studies". J Biomol Struct Dyn. 6 (2): 299–309. PMID 2482766.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Leslie AG, Arnott S, Chandrasekaran R, Ratliff RL (1980). "Polymorphism of DNA double helices". J. Mol. Biol. 143 (1): 49–72. PMID 7441761.
{{cite journal}}: CS1 maint: multiple names: authors list (link) - ↑ Wahl M, Sundaralingam M (1997). "Crystal structures of A-DNA duplexes". Biopolymers. 44 (1): 45–63. doi:10.1002/(SICI)1097-0282(1997)44:1. PMID 9097733.
- ↑ Lu XJ, Shakked Z, Olson WK (2000). "A-form conformational motifs in ligand-bound DNA structures". J. Mol. Biol. 300 (4): 819–40. doi:10.1006/jmbi.2000.3690. PMID 10891271.
{{cite journal}}: CS1 maint: multiple names: authors list (link)
